GSEA基本概念介绍
Gene Set Enrichment Analysis ,GSEA(基因集富集分析)用来评估一个预先定义的基因集的基因在与表型相关度排序的基因表中的分布趋势,从而判断其对表型的贡献。其输入数据包含两部分,一是已知功能的基因集,目标基因列表L (可以是GO注释、MsigDB的注释或其它符合格式的基因集定义),一是表达矩阵,功能基因列表S (也可以是排序好的列表),软件会对基因根据其与表型的关联度(可以理解为表达值的变化)从大到小排序,然后判断基因集内每条注释下的基因是否富集于表型相关度排序后基因表的上部或下部,从而判断此基因集内基因的协同变化对表型变化的影响。
这与GeneOntology,GO富集分析不同。GO富集分析是先筛选差异基因,再判断差异基因在哪些注释的通路存在富集;这涉及到阈值的设定,存在一定主观性并且只能用于表达变化较大的基因,即我们定义的显著差异基因。而GSEA则不局限于差异基因,从基因集的富集角度出发,理论上更容易囊括细微但协调性的变化对生物通路的影响,尤其是差异倍数不太大的基因集。GSEA分析不需要指定阈值(p值或FDR)来筛选差异基因,我们可以在没有经验存在的情况下分析我们感兴趣的基因集,而这个基因集不一定是显著差异表达的基因。GSEA分析可以将那些GO/KEGG富集分信息中容易遗漏掉的差异表达不显著却有着重要生物学意义的基因包含在内。
现在用文章里的常见的GSEA分析图来说明。
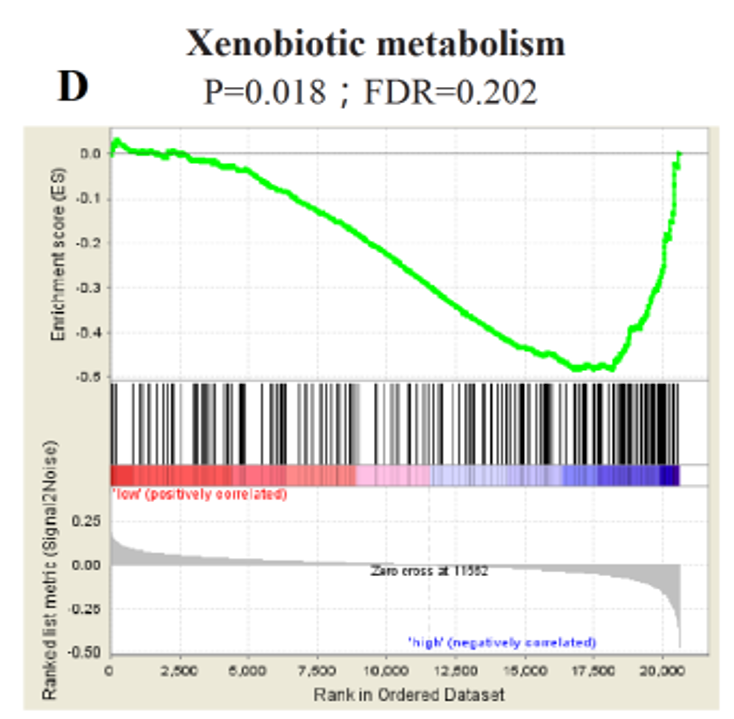
中间长的像条形码一样的竖线就是功能基因S在目标基因列表L中的位置。上面的绿线是Enrichment Score, ES, 富集分数。当功能基因S从上到下遍历目标基因列表L时,每遇到一个基因,ES分数就会增加,否则ES分数就会降低。
在实际工作中,主要用来分析某种处理是否会改变给定目标基因列表中基因的整体表达模式。
- See also
- GSEA结果解读
- GSEA学习笔记
- 如何实现GSEA-基因富集分析?